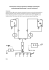Pobierz artykuł
Transkrypt
Pobierz artykuł
Zastosowanie metod interpolacyjnych do powiększania dwuwymiarowych obrazów medycznych Rafał Henryk Kartaszyński, Paweł Mikołajczak Wydział Matematyki, Fizyki i Informatyki UMCS w Lublinie Streszczenie: W artykule zajmujemy się problemem efektywnego powiększania obrazów medycznych, mającym istotne znaczenie we współczesnej medycynie. Proces ten wymaga zastosowania odpowiednich funkcji interpolacyjnych. Metody przedstawione w pierwszej części pracy są następnie analizowane. W końcowej dyskusji podjęto próbę wybrania najefektywniejszej metody pod względem jakościowym i czasu wykonania. Abstract: In this article, we deal with problem of effective enlarging of medical images, which has essential meaning in present medicine. This process requires suitable interpolation functions. Methods introduced in first part of this work are then analysed. In final discussion, attempt of selection of most effective method is taken, in respect of quality and runtime. Wstęp We współczesnej medycynie istotną rolę odgrywają aparaty diagnostyczne takie jak MRI, CT, PET i wiele innych. Pozwalają one lekarzom uzyskać zdjęcia mózgu, czaszki lub innych narządów. W niniejszej pracy skupiono się na zdjęciach głowy wykonanych metodą rezonansu magnetycznego i tomografii komputerowej. W wyniku badania takimi aparatami otrzymujemy przekroje głowy płaszczyznami prostopadłymi do osi ciała. Są to jednak obrazy o ograniczonej rozdzielczości, pozwalające obserwować ogólny wygląd struktury mózgu czy samej czaszki. Aby umożliwić lekarzom dokładniejszą analizę zdjęć konieczne jest ich powiększenie. Wymaga to dobrania odpowiedniej metody interpolacyjnej pozwalającej na jak najwierniejsze oddanie kształtu struktur przy jak największym powiększeniu. Idealny interpolator powinien również móc trafnie wyznaczyć brakujące pixele w realnym czasie. W obrazowaniu medycznym mamy do czynienia z różnymi formami zapisywania zdjęć. Należy podkreślić, że są one opisane w stopniach szarości. W zależności od producenta aparatu mogą to być pliki w standardzie DICOM, lub pliki zawierające dane o pixelu zapisane na ośmiu, dziesięciu czy dwunastu bitach. W niniejszej pracy wykorzystuje się obrazy składające się z pikseli przyjmujących wartości zapisywalne na dwóch bajtach. W pierwszej części artykułu przedstawione zostały różne metody interpolacji, poprzedzone opisem teoretycznym procesu interpolacji. Następnie przeprowadzono porównanie interpolatorów oraz zaprezentowano wyniki. Aby ocenić jakość poszczególnych metod przeprowadzono analizę statystyczną, wizualną, oraz czasu wykonania. Na podstawie otrzymanych danych wyprowadzono wnioski i zaproponowano najkorzystniejszą metodę interpolacji. Interpolacja Załóżmy, że chcemy obliczyć wartość funkcji, nie koniecznie dyskretną, w pewnym punkcie x Î R znając jej wartości w punktach dyskretnych. Wówczas wartość funkcji I zapisujemy jako: I ( x) = å I ( k ) × K ( x - k ) k gdzie K(x) jest ciągłą funkcją będącą pewnym jądrem interpolacji. Sumowanie odbywa się po k punktach sąsiadujących z punktem x, bez straty ogólności i dla ułatwienia zakładamy, że odległość pomiędzy kolejnymi k jest jednostkowa. Jest to ogólny wzór pozwalający wyznaczyć wartość pewnego jednowymiarowego dyskretnego sygnału w punkcie rzeczywistym. Ponieważ najczęściej wykorzystywane jądra są symetryczne i separowalne, ze względu na wymiary, przypadek sygnału dwuwymiarowego (2D) możemy przedstawić następująco: I ( x, y) = åå I (k , l ) × K 2 D ( x - k , y - l ) k l gdzie K 2 D ( x, y ) = K ( x) × K ( y ) , oraz analogicznie trój-wymiarowego (3D) I ( x, y, z ) = ååå I (k , l , m) × K 3D ( x - k , y - l , z - m) k l m gdzie K 3 D ( x, y, z ) = K ( x) × K ( y) × K ( z ) . W naszych rozważaniach sygnał I(k, l), czy I(k, l, m) będziemy rozumieli jako intensywność, w skali szarości, pixela obrazu w punkcie dyskretnym (k, l) dla obrazu 2D [1]. 50 Rys.1. przedstawia sposób wyznaczania wartości pixela w punkcie ( x, y ) na podstawie czterech sąsiadujących pixeli. Najpierw wyznaczane są wartości w punktach (x, ë y û) , (x, é y ù) , a następnie z nich interpolowana jest intensywność pixela w ( x, y ) . Gdy wyznaczamy punkt w przestrzeni trójwymiarowej postępujemy podobnie: w ww. sposób obliczamy wartość voxeli w płaszczyznach poniżej i powyżej szukanego punktu, a następnie interpolujemy wzdłuż osi OZ. Rys. 1. Jedno- wymiarowa dekompozycja dwu- wymiarowej interpolacji N x N punktu (x, y) Przegląd metod W naszych doświadczeniach badaliśmy wiele jąder interpolacyjnych: najbliższy sąsiad, liniowe, kubiczne, kosinusowe, Hermita (w wersji oryginalnej, James’a McCartney’a, Laurent’a de Soras), spline (w wersji sześciopunktowej, kubicznej, B – podstawowej, Catmull-Rom), oraz sinc (z oknami: prostokątnym, Blackman’a – Harris’a, Lanczos’a, Welch’a, Parzen’a, Hanna’a – Hamminga’a, Gaussa). Poniżej omówimy część metod. Interpolacja najbliższy sąsiad. Jest to najprostsza metoda interpolacji, wymaga jedynie N = 2 sąsiednich pixeli. Można zapisać jej jądro jako: ì1, K NN ( x ) = í î0, 0 £ x < 0.5 poza Jest to metoda bardzo niedokładna, ale szybka i najlepsza w prostych zastosowaniach. Interpolacja liniowa. [2] W naszych badaniach używaliśmy interpolacji liniowej dla dwóch sąsiednich punktów. ì1 - x , K L (x ) = í î0, 0 £ x <1 poza 51 Interpolacja kosinusowa. [4] Interpolacja opierająca się na funkcji trygonometrycznej cosinus, wykorzystująca dwa sąsiadujące punkty. ì (1 - cos( x × p )) ï , K C ( x ) = í1 2 ïî0, 0 £ x <1 poza Interpolacja kubiczna. [3] Jądro to opiera się na czterech punktach, oraz jest parametryzowane poprzez stałą α. Doświadczenia wykazały, że najbardziej optymalną i dającą pożądane wyniki niezależnie od interpolowanego obrazu jest stała α = – 1 / 2. ì(a + 2) x 3 + (a + 3) x 2 + 1, ï K Cub 4 ( x ) = ía x 3 - 5a x 2 + 8a x - 4a , ï î0, 0 £ x <1 1£ x < 2 poza Interpolacja spline Catmull-Rom. [7] Mając daną wartość pixela w czterech punktach P0, P1, P2, P3 wyznaczamy wartość w punkcie x Î R leżącym pomiędzy P1 a P2 w następujący sposób: [ I ( x ) = 0.5 × 1.0 x x2 2 0 0 ù é P0 ù é0 ê- 1 0 1 0 úú êê P1 úú 3 ê * x * ê 2 - 5 4 - 1ú ê P2 ú ê ú ê ú ë - 1 3 - 3 1 û ë P3 û ] Metoda ta często znajduje zastosowanie, prócz interpolacji obrazów, w wyznaczaniu łagodnej drogi pomiędzy punktami w symulacjach ruchu obiektów w przestrzeni 3D. Interpolacja sinc Jądro sinc: K sinc ( x ) = sinc ( x ) = sin (p × x ) p ×x jest idealnym iterpolatorem [1]. Ma jednak jedną wadę, nieskończoną dziedzinę. Dlatego w zastosowaniu praktycznym, przemnaża się ją przez odpowiednio dobraną funkcję – okno: ìï K ( x ) × w( x ) K Nsinc ( x ) = í sinc ïî 0 0£ x £ poza N 2 gdzie w(x) jest funkcją okna, a N jego szerokością. 52 Przedstawimy teraz kilka używanych przez nas okien. Okno Welch’a [10] ì æ x ö2 ï wwelch ( x, r ) = í1 - çè r ÷ø ï0 î x <r poza Okno Lanczos’a [8] ì æ xö ï sin ç p × r ÷ ø ï è wlanczos ( x, r ) = í x ï p× ï 0 r î x <r poza Okno Hann’a - Hamming’a [9][10] ì æ xö ïa + (1 - a ) × cosç p × ÷ whh ( x, r , a ) = í è rø ïî 0 x <r poza dla α = 1/2 jest to okno Hann’a, a dla α = 0.54 Hamming’a. Okno Hamming’a ma tę wadę, iż jest nieciągłe na brzegach, co prowadzi do widocznych artefaktów w obrazie. Są to metody najczęściej wykorzystywane do interpolacji danych. Wyznaczają wartość w punkcie w zależności od wartości różnej ilości jego sąsiadów. Najprostsze opierają się na dwóch, a bardziej zaawansowane (np. rodzina sinc) na praktycznie nieskończonej liczbie punktów sąsiednich. Co się tyczy funkcji sinc, doświadczenia wykazują, że najlepsze wyniki otrzymuje się dla jąder sinc o nieparzystej wielkości [6]. Ma ona jednak wadę – wykonuje się wolno, w porównaniu do metod prostszych, włącznie z metodą funkcji sklejanych (spline). Porównanie metod Interesuje nas jak najdokładniejsze zachowanie kształtu danej struktury przy powiększaniu fragmentów obrazów MRI i CT. Aby ocenić jakość różnych metod interpolacyjnych wybraliśmy jeden z przekrojów głowy pochodzących z Visible Human Project [11], o rozmiarze 512 x 512 pixeli. Został on następnie zmniejszony do obrazów o rozmiarach 448 x 448 (powiększenie x 1,1), 384 x 384 (x 1,3), 256 x 256 (x 2), 192 x 192 (x 2,6), 128 x 128 (x 4), 64 x 64 (x 8) pixeli. Następnie wykorzystując jedenaście metod interpolacyjnych powiększyliśmy je do rozmiaru oryginalnego. Oceny wyników dokonaliśmy poprzez ocenę różnicy natężeń odpowiadających sobie pixeli obrazu oryginalnego i wyinterpolowanego. Wybraliśmy tę metodę oceny gdyż interesuje nas efekt wizualny interpolacji – jak największe podobieństwo do oryginału. Dla wartości (0 - 65535) otrzymanych w wyniku operacji różnicy obliczyliśmy średnią różnicę w natężeniu pixeli oraz odchylenie standardowe od tejże średniej. Wyniki zamieszczone są w tabeli 1. i 2. oraz na odpowiednich wykresach. 53 Tabela 1. Średnia różnica w natężeniu pixeli obrazu oryginalnego i obrazów wyinterpolowanych. nearest Powiększenie neighbour linear cosine cubic Hermite Hermite Laurent de Soars spline spline CatmullRom sinc Lanczos sinc Welch sinc HannHamming X8 1495,990 1457,160 1350,750 1527,060 1354,230 1390,320 1390,010 1390,320 1484,180 1531,680 1440,050 X4 887,048 821,005 755,659 741,310 763,154 672,604 673,073 672,604 701,563 724,276 685,213 X2,6 624,923 556,259 475,839 456,631 477,353 417,732 419,493 417,732 409,743 426,028 403,276 X2 520,808 452,967 395,332 388,188 398,712 347,958 350,870 347,958 358,919 359,622 362,493 X1,3 271,880 242,353 201,201 278,938 203,331 227,504 229,690 227,504 262,570 246,475 273,681 X1,1 145,094 271,559 220,797 279,827 224,355 221,203 220,249 221,203 253,058 239,643 262,856 Rys. 2. Wykres średniej wartości różnicy natężenia odpowiadających sobie pixeli w zależności od rozmiaru powiększanego obrazu, dla różnych metod interpolacyjnych 54 Tabela 2. Odchylenie standardowe natężenia pixeli obrazu oryginalnego i obrazów wyinterpolowanych. Powięnearest kszenie neighbour linear cosine cubic Hermite Hermite Laurent de Soars spline spline CatmullRom sinc Lanczos sinc Welch sinc HannHamming X8 4083,780 3163,780 3233,160 3357,770 3223,180 3235,360 3254,750 3235,360 3320,230 3352,410 3288,060 X4 2534,850 1683,890 1706,780 1614,160 1699,550 1575,220 1584,380 1575,220 1565,360 1578,650 1560,280 X2,6 1729,330 1083,080 1114,480 941,169 1105,290 933,430 942,738 933,430 887,136 889,071 894,779 X2 1612,450 934,034 987,008 926,235 972,255 866,198 880,394 866,198 858,024 857,021 861,734 X1,3 1154,430 608,519 577,211 737,455 576,953 599,943 608,043 599,943 625,110 618,473 630,538 X1,1 808,182 588,010 492,952 737,734 500,739 581,136 580,478 581,136 610,855 605,158 615,988 Rys. 3. Wykres średniej wartości różnicy natężenia odpowiadających sobie pikseli w zależności od rozmiaru powiększanego obrazu, dla różnych metod interpolacyjnych 55 Średnie wartości różnic w natężeniu pikseli, dla dużych powiększeń (rzędu x8), wykazują mały wpływ wyboru metody na uzyskiwane wyniki. Błędy w tych wypadkach są duże, a jądra dają zbliżone wyniki (za wyjątkiem interpolacji - najbliższy sąsiad, która jest najmniej korzystna wizualnie). Dla powiększeń mniejszego rzędu (o większym zastosowaniu w medycynie) różnice między jądrami interpolacyjnymi są nieznacznie większe. Pozwalają Rys. 4. Porównanie czasów wykonania różnych metod interpolacyjnych [msec]. one jednak wyłonić grupę najlepszych interpolatorów, takich jak: rodzina funkcji spline (zwłaszcza Catmull-Rom), sinc. Analiza odchylenia standardowego pokazuje, że mimo, iż różnice średniej dla metody replikacji pikseli (nearest neighbour), w porównaniu z innymi metodami, są małe to wprowadza ona jednak większy zakres, w którym zmieniają się wartości różnicy. Wniosek ten (oraz prosta analiza wizualna obrazów Rys. 5.) wyklucza tę metodę z grona korzystnych interpolatorów dla powiększeń większych niż x1,1. Jednocześnie stwierdzamy, że najlepsze wyniki dają jądra sinc Hann’a-Hamming’a, Lanczos’a, czy spline Catmull-Rom. Interpolacja kosinusowa i Hermita dla małych powiększeń (x1,3 – 1,1) daje nawet lepsze wyniki. Na powyższym wykresie (Rys. 4) widzimy, iż interpolacje sinc są bardziej czasochłonne. Można próbować je bardziej optymalizować, ale kosztem większego zużycia pamięci operacyjnej (tablicowanie funkcji sinc). Nie przyniesie to jednak dużego zysku, ponieważ są to jądra dające najlepsze wyniki dla większej liczby sąsiadów (5 - 6), co stanowi główny powód tak długiego czasu wykonania. Chcąc wybrać metodę najbardziej optymalną, musimy zadać sobie pytanie, co jest ważniejsze: jakość powiększonego obrazu, czy szybkość wykonania. Biorąc pod uwagę czas wykonania, jak najwierniejsze powiększenie oraz ograniczając się do powiększeń znajdujących praktyczne zastosowanie (x2 – x8), należy uznać metodę Catmull-Rom za najlepszą przy powiększaniu. Jądra sinc, choć dają bardzo dobre wyniki, są wymagające obliczeniowo i ich obliczenie zajmuje najwięcej czasu. Catmull-Rom daje podobne wyniki do tej interpolacji, jest jednak mniej złożona obliczeniowo i wydaje się rozwiązaniem optymalny pod względem jakości jak i czasu wykonania. Jeżeli natomiast nie istotny jest czas w jakim uzyskamy wynik, najlepsze wyniki dają funkcje sinc, zwłaszcza z oknem Hann’a-Hamming’a lub Lanczos’a. Należy jednak 56 podkreślić, że z powodu na swojej nieskończonej natury jądro sinc powoduje powstawanie artefaktów w obrazie (mają one postać delikatnie zdwojonych krawędzi). Rys. 5a. Powiększenie x4 zdjęcia MRI metodą najbliższy sąsiad Rys. 5b. Powiększenie x4 zdjęcia MRI metodą liniowa Rys. 5c. Powiększenie x4 zdjęcia MRI metodą kosinusową Rys. 5d. Powiększenie x4 zdjęcia MRI metodą kubiczną 57 Podsumowanie Zaprezentowane metody wydają się być bardzo efektywnymi w powiększaniu obrazów medycznych. Interpolacje sinc (Lanczos, Hann-Hamming) oraz porównywalna z nimi spline Catmull-Rom, zachowują kształt, nie zniekształcają krawędzi i dają bardzo wyraźny powiększony obraz. Choć metoda testowania jest bardzo prosta i intuicyjna, wydaje się odpowiednia przy określaniu jakości wyinterpolowanych obrazów – największe zbliżenie do oryginału. Oczywiście można przeprowadzić analizę metodą MSE (Multiscale Entropy Analysis) [12], nie zmieni ona jednak ostatecznych wniosków i jest mniej intuicyjna. Interesujące wydaje się zastosowanie metod triangulacyjnych [13]. Interpolacja taka powinna lepiej odwzorowywać brzegi (kontury) struktur anatomicznych. Są to jednak algorytmy bardzo złożone i przez co kosztowne [14]. Zysk (w sensie wizualnym) z ich zastosowania może się okazać niewystarczający w porównaniu do czasu wykonania. Literatura 1. Thomas M. Lehmann, Claudia Gönner, Klaus Spitzer, “Survey: Interpolation Methods in Medical Image Processing”, IEEE Transactions on medical imaging, vol. 18, no. 11, November 1999 2. J. A. Parker, R. V. Kenyon, D. E. Troxel, „Comparision of interpolating methods for image resampling“, IEEE Trans. Med. Imag., vol. MI-2, pp. 31-39, 1983 3. P. E. Danielsson, M. Hammerin, „High accuracy rotation of images“, Computer Vision, Graphics and Image Processing, Vol.54, No. 4, pp.340-344, July 1992 4. K. G. Beauchamp, “Signal Processing”, George & Allen Unwin Ltd., 1973 5. Aili Li, Klaus Mueller, Thomas Ernst, “Methods for Efficient, High Quality Volume Resampling in the Frequency Domain”, IEEE Visualization, pp. 3-10, 2004 58